|
科普知识
|
科普文章
楔子初:2021年10月,命运突然交给我一个任务——给铠兰属建系统发育树。任务前提:没有足够的分子数据、没有经费可用来采样、疫情期间没有机会去国外出差、没有积累任何可用的形态数据、不能找人合作……用常规的方法构建系统发育树是不可能的,想要完成这个任务,只能另辟蹊径了。
铠兰属(Corybas Salisb.)是一个奇怪的属,属于兰科。它大约有170个物种,每一种都开着迷你、可爱的花,长着一片心形的叶子。从属的角度来看,它是广布的类群,从靠近南极的麦夸里岛一直到中国喜马拉雅山以北都有分布,主要分布于澳大利亚、新西兰、巴布亚新几内亚、印度尼西亚和马来西亚。然而,它几乎所有的成员物种都是地区特有的。如果你去GBIF网站上,把所有铠兰属照片都看一遍,你会发现不同地区的铠兰属植物长相是有规律的,甚至可以轻松地通过照片判断拍摄地! ▶澳大利亚的铠兰有三类:第一类圆滚滚,似糖球(C);第二类像个标准的弯月,或者像逗号(A);第三类像个小灯笼(B)。 ▶新西兰的铠兰有长长的“胡须”,圆脸盘,有些圆得很像第一类澳大利亚铠兰(D-G);新西兰还有两个种同澳大利亚小灯笼形的铠兰很像(B)。 ▶亚洲的铠兰长着奇怪又艳丽的脸,爱戴小红帽(H-J)。 ▶而澳大利亚到亚洲的岛链上那些铠兰或多或少都介于澳大利亚铠兰和亚洲铠兰之间,各种类型都有(A、H-J)。
如果我这样一个略微眼瞎的“植物盲”看照片都能一眼看出规律,那么我们可以利用形态来建树吗?根据我不太多的植物系统学知识,我知道通过花、叶、果的大小、颜色和毛被等形态性状可以用来建系统发育树。虽然我没办法从照片上量出花多大、叶子多大,但是本质上这些形态特征的数值只不过是为了定量,只要能定量地衡量形态,用别的方法不也行吗?比如说用机器学习中非常常见的图像处理! 专业做系统发育重建的植物学家可能会对此嗤之以鼻,因为形态特征建树容易受共衍征的影响,说人话就是:长得像的不一定是兄弟姐妹,可能只是碰巧。但是我们什么都没有,只有这些免费的照片,既然形态特征能建树,通过机器学习方法用照片建树不过是一种另类的“版本更新”而已。一筹莫展之下,能勉强建个树,数据来源还是免费的,这种好事,要啥自行车呀! 重点是:免费!免费!免费!不要998!不要98!不要钱!不需要采样!不需要测序!不需要央求师兄带你去出差!不需要学会爬树攀岩! 使用机器学习的图像识别方法来鉴定植物的案例不少见,比如说大名鼎鼎的形色、花伴侣等等APP,然而却没有人用这套方法进行植物系统发育树的构建。如果你都能知道照片里是啥植物了,你难道没法找出哪些植物的照片比较相似吗?唯一的不同之处在于:植物的识别是一种分类任务,也就是说,给模型一张图片,让模型判断这是A植物、B植物,还是C植物,而构建系统发育树是一个聚类任务,也就是给模型一堆图片,让模型把图片按照相似程度分堆。 那么我们怎么知道图片的相似程度呢?最简单的是:提取图片的特征值,计算图片之间特征值的距离,通过这个距离来衡量图片的相似程度,说起来比较抽象,但是如果你学过主成分分析,你可以想象一下:我们经常把两个主成分化成x和y轴,数据就分布在这个平面坐标系上,越是相似的数据就离得越近。一旦我们获得物种之间的距离,就能用距离来建树。 由于我太菜,我打算每一个步骤都用最简单的方式进行计算,用到的数学知识不能超过中学数学水平。比如说特征值,我果断放弃了自己建模,决定在优秀的预训练模型上进行微调(fine-tune,就像机器学习领域中所谓的“调参侠”那样),然后距离就用最简单的欧式距离吧(类似于中学时代学过的多维直角坐标系下两点间距离的计算公式)。建树也先别整啥K-mean、最大似然法了,我们先用最简单的层次聚类法,就是说每次把最相似的两个物种聚到一起,聚完合并当一个物种看待,下一轮又把最相似的两个聚到一起,不停地重复,最后就只剩下两堆物种,自然而然就建完了。至于图像处理领域的“图像增强”方法,包括图像旋转、切片、滑动窗口等等方法都先不考虑。 头疼的是,我们怎么知道我们建的树是否靠谱呢?聚类是一种无监督学习,也就是没有“标准答案”,但是聚类结果的好坏还是可以评估的,比如说:每一次将两堆物种进行聚类的时候都可以用轮廓分数(Simplified Silhouette Index,SSI)来评估聚类的可靠程度。轮廓系数的取值范围是[-1, 1],数字越大越好。 当然啦,整个过程中最好对数据进行抽样,这样可以产生一大堆树,方便进行效果评估。比如说我们建出1000棵树,使用对称差(Symmetric Difference)算出两两之间的差异,如果每一棵树差异都很大,那说明结果的偶然性很强,所以同一模型建出来的1000棵树之间对称差越小越好。然后我们用PAUP*把这1000棵树合并成一棵50%多数一致树;同时我们随机产生5000棵树,合并成一棵50%多数一致随机树。然后,我们可以使用对称差算出我们的树和随机树之间的差异。如果我们建的树是可靠的,我们的树至少要和随机树不一样吧?因此和随机树之间的对称差应当越大越好。 说起来,我此前还没有玩过图像数据,每天都面对着开不出花来的数字,这是多好的机会呀!感觉Python图标都开始可爱了起来!废话不多说,准备好图像和Python,用你心爱的Python IDE打开文章附件2和附件3,跟着我开始玩数据吧! 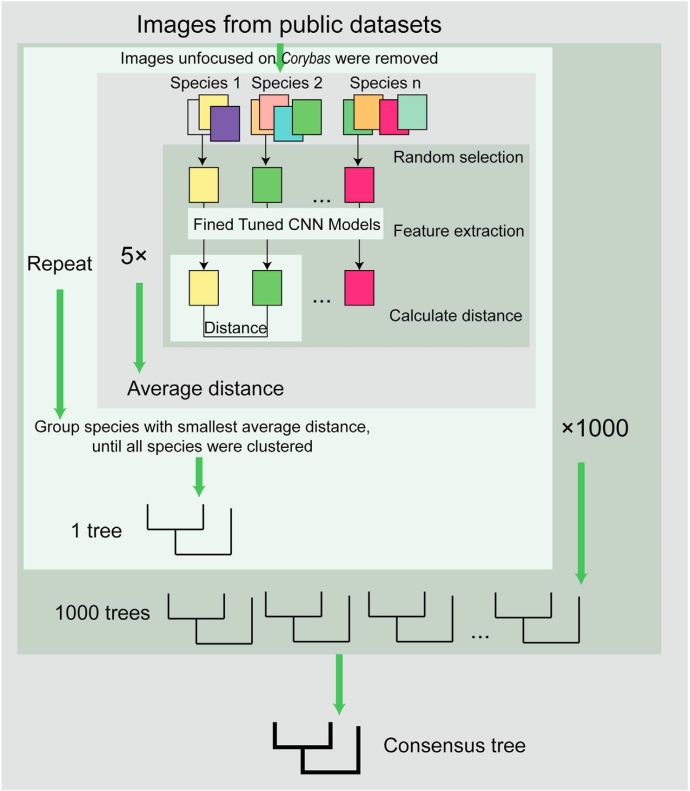 图像聚类流程图 第一步:下载图片 如果你真的一无所有,连照片也找不出来,有几个方法可以获得数据:①GBIF(https://www.gbif.org/),此数据库有全球植物图像,量大,图片几乎都可以免费公开使用,但是照片质量良莠不齐。GBIF官方提供了API,支持爬虫,下载照片的时候最好确认一下图片的知识共享协议,也就是说CC协议;②PPBC(http://ppbc.iplant.cn/),中国植物图像库,网站不允许爬虫,有版权;③其他,比如说文献里的照片,不同文章有不同的协议,需要去查一下文章的CC协议。
第二步:预训练模型的微调 你可以自己选择预训练图像处理模型,我选择了ResNet50和InceptionResNetV2,因为这俩都是很常用、口碑还不错的模型。我用具有超过100张照片的物种来对预训练模型进行微调。先把预训练模型的全连接层(fully connected layer)都丢掉,把ResNet50的最后5层以及InceptionResNetV2的最后281层设置成可训练(你可以自己决定训练哪些层,也可以尝试设置不同的可训练层,找出最好的那个“配方”)。训练完成后,将微调好的模型参数保存起来待用。
第三步:提取图像特征值并进行聚类 分别用没有微调的ResNet50、没有微调的InceptionResNetV2、微调好的ResNet50、微调好的InceptionResNetV2模型对每个物种的图像提取图像特征。我们使用的是层次聚类法,每一次只能把距离最短的两个物种堆聚到一起,所以我们需要算出哪两堆物种的图像距离最短。计算这个距离的时候,每对物种堆都要单独计算。比如说我要知道物种堆A和物种堆B之间的距离,我先从A的照片里随机抽一张出来,提取特征,然后从B的照片里随机抽一张出来,提取特征,算出特征值之间的差异,即距离,这个步骤重复5遍,得到5个距离。我们把这5个距离取平均值当作最后的距离值(这个距离值还可以当作树的支长)。我们简单排个序,把距离值最小的两堆物种合并起来,把这次合并的过程记录下来。不停地重复以上步骤,直到最后只剩下两堆物种,此时我们就可以顺利得到一棵树。重复建树的过程1000次,就得到1000棵树。我们把这1000棵树写成nexus文件(nexus文件本质是一种文本文件,其格式很简单,你们找一个nexus文件盯着看会儿就懂了),这个文件可以用建树的软件打开,就能看到树了。具体操作方法就是打开文章的附件2,修改路径和必要参数,比如说重复次数等,然后运行Python脚本即可,如果遇到报错,先自行debug,解决不了的话可以发邮件和我讨论。
在这上图中,我们只用了图片数量≥5的物种,括号里是物种分布区域和图片数量。可以看出来,大部分分支符合我们预期,比如说新西兰的物种倾向于聚在一起,亚洲的物种倾向于聚在一起,澳大利亚的物种在散在多个分支中。但是有些节点没有解决,还有些节点不符合我们的预期。比如说很明显,当模型遇到灯笼形状花朵的铠兰时,它容易同月牙形混淆;最先分支出来的C. hipidus和C. walliae的位置似乎不太正确。所以说,我们的方法有不完美的地方。 我建议,如果你想研究的类群有一些物种存在分子数据,可以先用分子数据建一个骨架树,将图像聚类的结果填充进骨架树中,补充没有分子数据的类群之间的关系,比如说你可以写一个惩罚函数,惩罚那些同分子骨架树矛盾的图像聚类树。 第四步:模型对比和聚类效果评价 轮廓分数SSI怎么得到呢?首先要把物种和节点信息整理成表格,参照附件4的第2-19张sheet,格式看上去有点晦涩,实则很简单。第一列是物种名,第二列开始是节点信息。打个比方,fir50对应的是上面这张图,Node.1就是第一个分支,我们从图上可以看出来,除了C. hispidus之外,其他都聚在一起,那么C. hispidus的在第一个节点的编号就是0,其他全都是1,由于C. hispidus后面没有分支了,所以C. hispidus只有第一列有数据,后面就没了。Node.2就是接下来的节点,即C. walliae同别的物种分开的那个节点,在这个节点上,C. walliae就是0,其他物种就是1;如果遇到一个节点后面发散出多个分支,那么就依次编号0、1、2、3…第一个分支线后面连着的那些物种就都是0,第二个分支线后面连着的那些物种就都是1,依此类推。整理完分支数据之后,把数据输出成csv,把csv文件输入到附件3即可,算完之后就是附件4的SSI中的结果。如果遇到Python脚本报错,需要核对下你的csv文件是不是编码不正确,比如说是否是UTF-8无签名的。Linux系统还需要注意转化换行符和文件路径里的斜杠。 至于对称差,其实是PAUP*算出来的,具体去看一下PAUP*的教程,按教程操作。 结果就是:微调版InceptionResNetV2模型建的树效果最好。这说明预训练模型的选择对建树效果有影响,需要多尝试不同的预训练模型。 图像聚类的优点: ①免费,可用的图片数据量会逐年增加; ②虽然是使用形态特征做系统发育的一种“更新”,但是由于深度学习网络层数非常多,每一层都可以学到图像的某一方面信息,因此可以充分利用图片的各种信息,或许会比直接人为测量形态特征更加客观,对植物形态信息的利用也更加丰富。 ③在分子数据不足的时候,或许可以补充分子数据缺失类群的系统发育位置。 存在的问题: ①我们的图像数据量太小了,如果图多点,效果可能会更好,但是尽管少,居然还是能建成模型,这是有赖于“巨人的肩膀”——预训练模型。 ②不同类群的情况不同,很多兰科植物形态和分子建成系统发育关系比较吻合,用图像聚类可能会获得比较好的结果,但是其他植物类群就不好说了,因此需要尝试更多类群。 ③代码有很多优化空间,比如说特征值提取的部分是可以写成多线程的,我没有写是因为我的电脑算力足够。当任务量大的时候需要对代码进行更多优化,图片多、物种多的时候,内存管理也需要优化。 ④这套方法实在是太便宜了,效果不算完美,但是大致可靠,用了不亏,如果希望变得更可靠点,可以同分子数据联合建树,还可以在模型优化上花点功夫,比如说我们没有用任何图像增强方法,以后可以试试看;聚类效果评估还有很大的探索空间,比如说当对比随机一致树和我们的一致树的对称差时,我们对比的是两棵树,而当对比每个模型建成的1000棵树的对称差时,我们对比的是1000棵树,两个对称差之间没法进行对比。 ⑤文本只是初探,未来道阻且长。各位“植物人”们!不要只蹲在“植物星球”的深坑里了,不妨睁眼看看世界!看看其他领域都在做什么,你会发现世界辽阔、星辰璀璨、大有可为!
总结: 看到这儿,你是不是觉得这套方法很简单,迫不及待想去玩一下,甚至还想DIY一些新东西?快去试试看吧!反正花不了多少电费!祝大家玩得开心! |
版权所有 Copyright © 2002-2016 中科院昆明植物研究所,All Rights Reserved 【滇ICP备05000394号】
地址:中国云南省昆明市蓝黑路132号 邮政编码:650201
点击这里联系我们